Tanon 5200 全自动化学发光图像分析系统

*如果您在实验中遇到任何问题, 请联系我们, 我们将为您提供帮助.
联系电话: 010-5158 2336转803 电子邮箱: help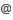 yph-bio.com
yph-bio.com
联系电话: 010-5158 2336转803 电子邮箱: help
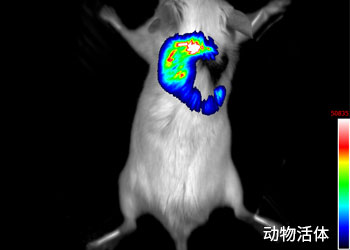
Tanon 5200发光成像系统
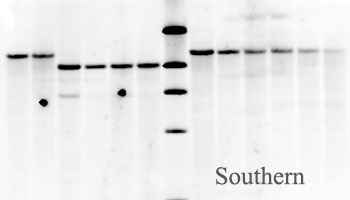
Tanon 5200是专业的化学发光图像分析系统,可对ECL发光等直接成像,获得实验结果。以往我们主要使用暗室曝光来进行ECL检测,但是建造暗室浪费空间,在暗室中曝光步骤繁琐,冲洗X胶片时会接触到有毒的化学试剂。现在使用发光成像系统进行ECl成像已经逐渐取代传统的暗室曝光检测。

性能特点
- 高分辨率:2750×2200分辨率,原生605万像素,16bit图像,保证成像效果
- 深度制冷:使用Peltier技术对CCD进行深度制冷,可以进行长时间的ECL发光成像
- 像素合并:通过像素合并,进一步提高ECL成像的灵敏度,捕获微弱的发光信号
- 大光圈定焦镜头:使用F0.80的大光圈定焦镜头,通光能力比F0.95镜头提高40%
- 样品台可选:标配抽提式双载物对焦平台,可适应不同高度样品
- 全中文软件:自主开发,全中文拍摄、分析软件,操作简便,终身升级
 |
 |
 |
 |
 |
 |
技术规格
- 摄像头: 美国原装 FLI品牌高分辨率低照度数码制冷相机
- 感光芯片: CCD芯片:Sony ICX694
- 冷却方式: 半导体制冷
- 冷却温度: 低于环境温度65℃(绝对温度-40℃,动态实时显示CCD制冷温度)
- 感光效率: CCD芯片光电转换效率:High QE: ≥78%
- 暗电流: <0.001 e-/pixel/sec.
- 读出燥声: 3e- RMS
- 有效像数: 2750×2200
- 像数密度 : 16 bit (0 - 65535色)
- 像素合并: 1×1,2×2,3×3,4×4,5×5
- 分辨率: 605万像素
- 动态范围: ﹥4.6个数量级
- 电动镜头: F0.80大光圈定焦镜头,通光量比F0.95镜头提高40%;
- 变 焦: 标配抽屉式双位载物对焦平台,可兼容拍摄样品厚度0.01mm~10cm
- 照明模式: 透射白光,反射白光
- 激发光源: 透射:LED白光板; 双侧反射:反射白光灯(冷光)
- 拍摄面积: 15×15cm
软件特点
- 全中文软件,自动识别8bit、10bit、14bit、16bit的图像以及序列图像
- 具有实时图像采集软件,可用于核酸、蛋白电泳凝胶图像和化学发光图像的采集
- 具有自动曝光功能,可以实时自动曝光
- 单次成像:具有长时间曝光功能,可实现单张画面长时间曝光
- 累积曝光多次成像功能:可以在很长曝光时间内多次成像,且每次成像的曝光时间可以累积,从而避免反复曝光,而且用户可以挑选中意的图像保存。
- 具有序列图像保存功能,无需单张图片分别存储
- 在拍摄中可显示过饱和像素,保证精确定量
- 具有加注功能,可添加各种格式的文字注释或符号
- 具有分析软件,可进行自动条带检测,自动分子量测算,自动条带浓度测算,相对含量百分数分析,绝对浓度、密度计算
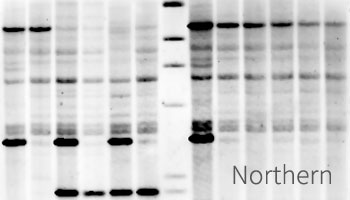
- 具有点杂交和菌落计数分析功能,可进行菌落克隆计数、菌落面积分析与排序
- 图像叠加分析功能:可对两个图像进行合并显示,并进行分析
- 图像输出格式:8bit Tif、16bit Tif、bmp、jpg、序列图像
应用发表文献
- Guo X, Wang L, Li J, Xu Y H, et al. Structural insight into autoinhibition and histone H3-induced activation of DNMT3A[J]. Nature, 2014.
- Chu B B, Liao Y C, Qi W, Song B L,et al. Cholesterol Transport through Lysosome-Peroxisome Membrane Contacts[J]. Cell, 2015, 161(2): 291-306.
- Li W Y, Ma M D, Feng Y, Guo H W,et al. EIN2-Directed Translational Regulation of Ethylene Signaling in Arabidopsis[J]. Cell, 2015
- Cui X, Lu F, Qiu Q, Cao X F,et al. REF6 recognizes a specific DNA sequence to demethylate H3K27me3 and regulate organ boundary formation in Arabidopsis[J]. Nature Genetics, 2016
- Xie C, Zhang Y P, Song L, et al. Genome editing with CRISPR/Cas9 in postnatal mice corrects PRKAG2 cardiac syndrome[J]. Cell Research, 2016.
- Song W, Liu L, Wang J, et al. Signature motif-guided identification of receptors for peptide hormones essential for root meristem growth[J]. Cell Research, 2016.
- Chen F, Di H, Wang Y, et al. Small-molecule targeting of a diapophytoene desaturase inhibits S. aureus virulence[J]. Nature chemical biology, 2016
- Lou Y, Xu X F, Zhu J, et al. The tapetal AHL family protein TEK determines nexine formation in the pollen wall[J]. Nature communications, 2014, 5.
- Jian Fang, Jingdong Cheng, Jiaolong Wang, et al. Hemi-methylated DNA opens a closed conformation of UHRF1 to facilitate its histone recognition[J]. Nature Communications, 2016
- Xu F, Zhao H, Feng X, et al. Single‐Cell Chemical Proteomics with an Activity‐Based Probe: Identification of Low‐Copy Membrane Proteins on Primary Neurons[J]. Angewandte Chemie, 2014, 126(26): 6848-6851.
- Zhang Z, Zheng X, Yang J, et al. Maize endosperm-specific transcription factors O2 and PBF network the regulation of protein and starch synthesis[J]. Proceedings of the National Academy of Sciences (PNAS), 2016
- Liu H, Shi J, Sun C, et al. Gene duplication confers enhanced expression of 27-kDa γ-zein for endosperm modification in quality protein maize[J]. Proceedings of the National Academy of Sciences (PNAS), 2016
- Xu H, Lin Z, Li F, et al. Dimerization of elongator protein 1 is essential for Elongator complex assembly[J]. Proceedings of the National Academy of Sciences (PNAS), 2015: 201502597.
- Zhang Z, Yang J, Wu Y. Transcriptional Regulation of Zein Gene Expression in Maize through the Additive and Synergistic Action of opaque2, Prolamine-Box Binding Factor, and O2 Heterodimerizing Proteins[J]. Plant Cell, 2015, 27(4): 1162-1172.
- Yang Y, Fu D, Zhu C, et al. The RING-Finger Ubiquitin Ligase HAF1 Mediates Heading date 1 Degradation during Photoperiodic Flowering in Rice[J]. Plant Cell, 2015: tpc. 15.00320.
部分用户
|
|
|
|
咨询留言


